[dataset] Sleep-edf
2020. 9. 14. 22:10ㆍPapers/Sleep Stage
Dataset Sleep-edf 정리
dataset 다운로드 url : https://physionet.org/content/sleep-edfx/1.0.0/
- 위의 링크를 통해서 2013 + 2018 dataset을 다운받을 수 있습니다.
- 추가적으로 2013데이터셋만 사용하실 분들은 첨부된 sh파일을 참고하시면 될 것 같습니다.
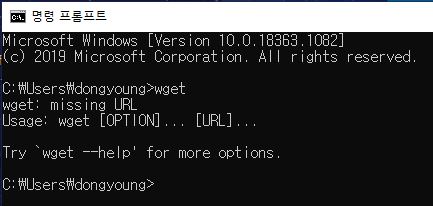
- windows 환경에서 다운 받고 사용할 때 wget설치가 필요합니다. 아래의 파일을 다운(64bit 운영체제 기준)받은 후 windows/system32/ 에 다운받아 사용하시면 cmd를 통해 wget이 정상적으로 동작하는 지를 확인할 수 있습니다.
- 위의 그림과 같이 cmd에서 wget을 쳤을 때 다음과 같은 상황이 나오면 sh파일을 통해서 설치가 가능합니다.
데이터셋의 경우 edf파일로 구성이 되어있기 때문에, python을 통해서 해당 파일을 열기 위해 pyEDFlib을 설치해야 합니다.
- pip install pyEDFlib
import numpy as np
from pyedflib import highlevel
import matplotlib.pyplot as plt
import os
import pandas as pd
import random
import shutil사용할 라이브러리 import 과정
shutil : move copy 등 사용하기 위한 라이브러리
os : makdirs & listdir 같이 폴더 생성 및 폴더 안에 있는 파일 탐색을 위한 라이브러리
matplotlib : 그림을 그리기 위한 라이브러리
numpy : 데이터를 활용하기 위한 라이브러리
etc
def search_annotations_edf(dirname): # annotations파일 이름이 Hyponogram.edf이기 때문에 해당 파일을 찾기 위한 함수
filenames = os.listdir(dirname)
filenames = [file for file in filenames if file.endswith("Hypnogram.edf")]
return filenames
def search_signals_edf(dirname): # signals 파일 이름이 PSG.edf이기 때문에 해당 파일을 찾기 위한 함수
filenames = os.listdir(dirname)
filenames = [file for file in filenames if file.endswith("PSG.edf")]
return filenames
def search_correct_annotations(dirname,filename): # signals파일에 대한 annotations을 저장하고 있는 파일을 찾기 위한 함수
search_filename = filename.split('-')[0][:-2]
file_list = os.listdir(dirname)
filename = [file for file in file_list if search_filename in file if file.endswith("Hypnogram.edf")]
return filename
# npy형태로 변환 후 사용할 메소드들
def search_signals_npy(dirname):
filenames = os.listdir(dirname)
filenames = [file for file in filenames if file.endswith(".npy")]
return filenames
def search_correct_annotations_npy(dirname,filename):
search_filename = filename.split('-')[0][:-2]
file_list = os.listdir(dirname)
filename = [file for file in file_list if search_filename in file if file.endswith("npy")]
return filename
def search_correct_signals_npy(dirname,filename):
search_filename = filename.split('-')[0][:-2]
file_list = os.listdir(dirname)
filename = [file for file in file_list if search_filename in file if file.endswith("npy")]
return filename아래 소스들에서 사용할 함수 선언
path = 'D:/dataset/data_2013/' # sleep-edf 2013 데이터를 가지고 있는 폴더 명
annotations_edf_list = search_annotations_edf(path)
signals_edf_list = search_signals_edf(path)
print('signals edf file list')
print(signals_edf_list)
print('annotations edf file list')
print(annotations_edf_list)
annotations 파일과 signals 파일을 확인
for filename in signals_edf_list:
print('signals file name : %s , annotations file name : %s'%(filename,search_correct_annotations(signal_path,filename)[0]))signals와 같은 annotations파일을 찾기 위한 반복문 ( 해당 2013 dataset을 설치를 하게 되면 하나의 파일이 signals 와 annotations으로 구성되어 있지 않고 사용할 수 없게 하나의 파일로만 구성되어 있기 때문에 해당 반복문을 통해서 있는 파일을 확인함. )
epoch_size = 30
sample_rate = 100
save_signals_path = path + 'origin_npy/'
save_annotations_path = save_signals_path+'annotations/'
os.makedirs(save_annotations_path,exist_ok=True)
os.makedirs(save_signals_path,exist_ok=True)
for filename in signals_edf_list:
signals_filename = filename
annotations_filename = search_correct_annotations(signal_path,filename)[0]
signals_filename = path + signals_filename
annotations_filename = path + annotations_filename
_, _, annotations_header = highlevel.read_edf(annotations_filename)
label = []
for ann in annotations_header['annotations']:
start = ann[0]
length = ann[1]
length = int(str(length)[2:-1]) // epoch_size # label은 30초 간격으로 사용할 것이기 때문에 30으로 나눈 값이 해당 sleep stage가 반복된 횟수이다.
if ann[2] == 'Sleep stage W':
for time in range(length):
label.append(0)
elif ann[2] == 'Sleep stage 1':
for time in range(length):
label.append(1)
elif ann[2] == 'Sleep stage 2':
for time in range(length):
label.append(2)
elif ann[2] == 'Sleep stage 3':
for time in range(length):
label.append(3)
elif ann[2] == 'Sleep stage 4':
for time in range(length):
label.append(3)
elif ann[2] == 'Sleep stage R':
for time in range(length):
label.append(4)
else:
for time in range(length):
label.append(5)
label = np.array(label)
signals, _, signals_header = highlevel.read_edf(signals_filename)
signals_len = len(signals[0]) // sample_rate // epoch_size
annotations_len = len(label)
if signals_header['startdate'] == annotations_header['startdate']:
print("%s file's signal & annotations start time is same"%signals_filename.split('/')[-1])
if signals_len > annotations_len :
signals = signals[:3][:annotations_len]
elif signals_len < annotations_len :
signals = signals[:3]
label = label[:signals_len]
else:
signals = signals[:3]
signals = np.array(signals)
np.save(save_signals_path + signals_filename.split('/')[-1].split('.')[0],signals)
np.save(save_annotations_path + annotations_filename.split('/')[-1].split('.')[0],label)
if (len(signals[0])//sample_rate//epoch_size != len(label)):
print('signals len : %d / annotations len : %d'%(len(signals[0])//sample_rate//epoch_size,len(label)))
else:
print("%s file''s signal & annotations start time is different"%signals_filename.split('/')[-1])
annotations은 총 6개의 class로 구성되어 있다. 0~5부터 순차적으로 Wake / N1 / N2 / N3 / REM / ??? 으로 구성이 되어 있는데 ???의 경우 어떠한 클래스에도 속하지 않는 케이스를 의미하며 N1 ~ N3는 3일수록 깊은 잠을 의미하고 N1에 가까운 수면은 얕은 잠을 의미한다.
해당 signals와 annotations을 npy 파일 형태로 변환한다 . ( numpy )
epoch_size = 30
sample_rate = 100
path = 'D:/dataset/data_2013/origin_npy/'
signals_npy_list = search_signals_npy(path)
print(signals_npy_list)
channel_name_list = ['Fpz-Cz/','Pz-Oz/','EOG/']
for channel_index,channel_name in enumerate(channel_name_list):
save_path = path + channel_name
os.makedirs(save_path,exist_ok=True)
for filename in signals_npy_list:
signals_filename = filename
signals_filename = path + signals_filename
signals = np.load(signals_filename)
signals = signals[channel_index].reshape(1,-1)
print(signals.shape)
np.save(save_path + filename,signals)위의 반복문을 통해서 채널별로 signals를 분리한다. 실제로 우리가 사용할 signals은 Fpz-Cz 채널이다. (EEG)
epoch_size = 30
sample_rate = 100
path = 'D:/dataset/data_2013/origin_npy/Fpz-Cz/'
annotations_path = 'D:/dataset/data_2013/origin_npy/annotations/'
signals_npy_list = search_signals_npy(path)
print(signals_npy_list)
for filename in signals_npy_list:
signals_filename = path + filename
annotations_filename = annotations_path+search_correct_annotations_npy(annotations_path,filename)[0]
signals = np.load(signals_filename)
label = np.load(annotations_filename)
if len(signals[0])//sample_rate//epoch_size != len(label):
print('%s is fault'%filename)
저장한 파일의 길이가 signals 와 label의 길이가 같은지를 확인하는 작업이다.
fs = 100 # Sampling rate (100 Hz)
epoch_size = 30
path = 'D:/dataset/data_2013/origin_npy/annotations/'
annotations_npy_list = search_signals_npy(path)
check_index_size = 10
for filename in annotations_npy_list:
label_info = np.zeros([5],dtype=int)
label = np.load(path + filename)
plt.plot(label)
plt.show()
print('='*20)
fs = 100
epoch_size = 30
#data = np.random.uniform(0, 100, 1024)
path = 'D:/dataset/data_2013/origin_npy/annotations/'
signals_path = 'D:/dataset/data_2013/origin_npy/Fpz-Cz/'
save_annotations_path = path + 'remove_wake/'
save_signals_path = signals_path + 'remove_wake/'
os.makedirs(save_annotations_path,exist_ok=True)
os.makedirs(save_signals_path,exist_ok=True)
annotations_npy_list = search_signals_npy(path)
check_index_size = 10
total_label = np.zeros([6],dtype=int)
for filename in annotations_npy_list:
label = np.load(path + filename)
signals_filename = search_correct_signals_npy(signals_path,filename)[0]
signals = np.load(signals_path+signals_filename)
for remove_start_index in range(0,len(label),1):
#print(np.bincount(label[remove_start_index:(remove_start_index+check_index_size)],minlength=6)[0])
if(np.bincount(label[remove_start_index:(remove_start_index+check_index_size)],minlength=6)[0] != check_index_size):
break
for remove_end_index in range(len(label),-1,-1,):
#print(np.bincount(label[remove_end_index-check_index_size:(remove_end_index)],minlength=6)[0])
if(np.bincount(label[remove_end_index-check_index_size:(remove_end_index)],minlength=6)[0] != check_index_size and np.bincount(label[remove_end_index-check_index_size:(remove_end_index)],minlength=6)[5] == 0 ):
break
#print('remove start index : %d / remove end index : %d'%(remove_start_index,remove_end_index))
label = label[remove_start_index:remove_end_index+1]
signals = signals[0,remove_start_index*fs*epoch_size:(remove_end_index+1)*fs*epoch_size].reshape(1,-1)
#print(np.bincount(label,minlength=6))
if len(label) ==len(signals[0])//30//fs:
np.save(save_annotations_path+filename.split('.')[0],label)
np.save(save_signals_path+signals_filename.split('.')[0],signals)
for i in range(6):
total_label[i] += np.bincount(label,minlength=6)[i]
print(total_label)
label을 확인하면 Wake와 ???의 수가 시작과 끝쪽에 비정상적으로 쏠린 모습을 확인할 수 있다. 해당 부분을 해결하기 위해서 ( Wake 와 ???의 수를 줄이기 위해 ) 인덱싱을 제한해 주는 작업을 수행한다. slide window 방식으로 한칸씩 이동하면서 label이 Wake나 ???로 이루어져 있을 경우 제거하면서 최종적으로 Wake 또는 ???이 아닌 class가 등장하면 해당 label의 인덱싱을 통해 signals의 크기와 일치시켜주는 작업을 수행한다.
해당 인덱싱을 통해 라벨링을 맞춰주면 클래스의 수는 다음과 같을 것이다.
4258 / 2762 / 17340 / 5575 / 7522 / 59
여기서 class의 수가 논문에 기재된 수와 다른 이유는 하나의 환자 파일이 정상적이지 않은 것과 Wake를 구별하는 시작 위치와 끝 위치 (remove_start_index / remove_end_index )가 일치하지 않기 때문이다. 해당 부분을 정확하게 맞추기 위해서는 실험을 통해 알아봐야 할 것이다. ( 유사한 정도만 만들고 실험하여 결과를 내기 위해서 똑같이 만들지는 않음 )
fs = 100
epoch_size = 30
#data = np.random.uniform(0, 100, 1024)
path = 'D:/dataset/data_2013/origin_npy/annotations/remove_wake/'
signals_path = 'D:/dataset/data_2013/origin_npy/Fpz-Cz/remove_wake/'
annotations_npy_list = search_signals_npy(path)
total_label = np.zeros([6],dtype=int)
for filename in annotations_npy_list:
label = np.load(path + filename)
signals_filename = search_correct_signals_npy(signals_path,filename)[0]
signals = np.load(signals_path+signals_filename)
#print('remove start index : %d / remove end index : %d'%(remove_start_index,remove_end_index))
#print(np.bincount(label,minlength=6))
if len(label) !=len(signals[0])//30//fs:
print('file is fault!!!')
for i in range(6):
total_label[i] += np.bincount(label,minlength=6)[i]
print(total_label)
x = np.arange(len(total_label))
plt.bar(x,total_label,width=0.7)위의 코드를 통해서 라벨링 상태를 확인하게 되면 아래와 같이 나온다.
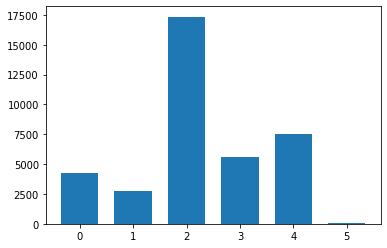
위의 그림을 보면 알 수 있듯이, N2의 수는 굉장히 많음에 비해서 그 외의 수는 적은 편임을 알 수 있다.
해당 부분에 있어서 class imbalanced 문제가 발생할 소지가 충분함. ( 하지만 N1의 경우 class의 수가 많더라도 성능이 나쁜 편임 -> 모델 또는 사람이 구별하는 것 또한 어려운 부분 )
이와 같이 해당 edf 데이터셋을 npy형태로 변환하여 사용하기 쉽게 만들고, 추가적으로 wake와 ??? 클래스들을 최대한 제거함으로써 사용하기 좋게 변환하였다. 다음 포스팅을 통해서 해당 데이터셋을 활용하여 학습을 하는것에 대해서 다룰 예정이다.
해당 주피터노트북 파일은 다음을 통해 확인할 수 있습니다.